Ảnh hưởng của hiện tượng mất phần tử trung hòa trong phân tích hệ protein dựa trên khối phổ với phương pháp thu nhận dữ liệu độc lập
Trong nghiên cứu sinh học phân tử và y học, việc xác định peptide và protein từ mẫu sinh học là bước then chốt để hiểu rõ các quá trình sinh lý và bệnh lý. Đặc biệt, các phosphopeptides - sản phẩm của quá trình phosphoryl hóa protein đóng vai trò quan trọng trong điều hòa hoạt động tế bào và đã trở thành trọng tâm trong nhiều nghiên cứu. Do đó, việc phát triển các phương pháp xác định phosphopeptides là rất cần thiết. Một trong những phương pháp rất mạnh được sử dụng rất nhiều trong các nghiên cứu là phân tích khối phổ (mass spectrometry - MS) - một phương pháp phổ biến để xác định peptide và protein trong các mẫu sinh học. Data-independent acquisition (DIA) là phương pháp phân tích khối phổ hiện đại và chứng minh được độ hiệu quả cao. Một thách thức lớn trong phân tích DIA là hiện tượng mất phần tử trung hòa (neutral loss). Đặc biệt đối với phospho- peptide, việc mất phần tử H3PO4) có thể gây ảnh hưởng đến độ chính xác trong nhận diện peptide. Nghiên cứu này sẽ tập trung vào đánh giá tác động của neutral loss này đối với quá trình phân tích DIA.
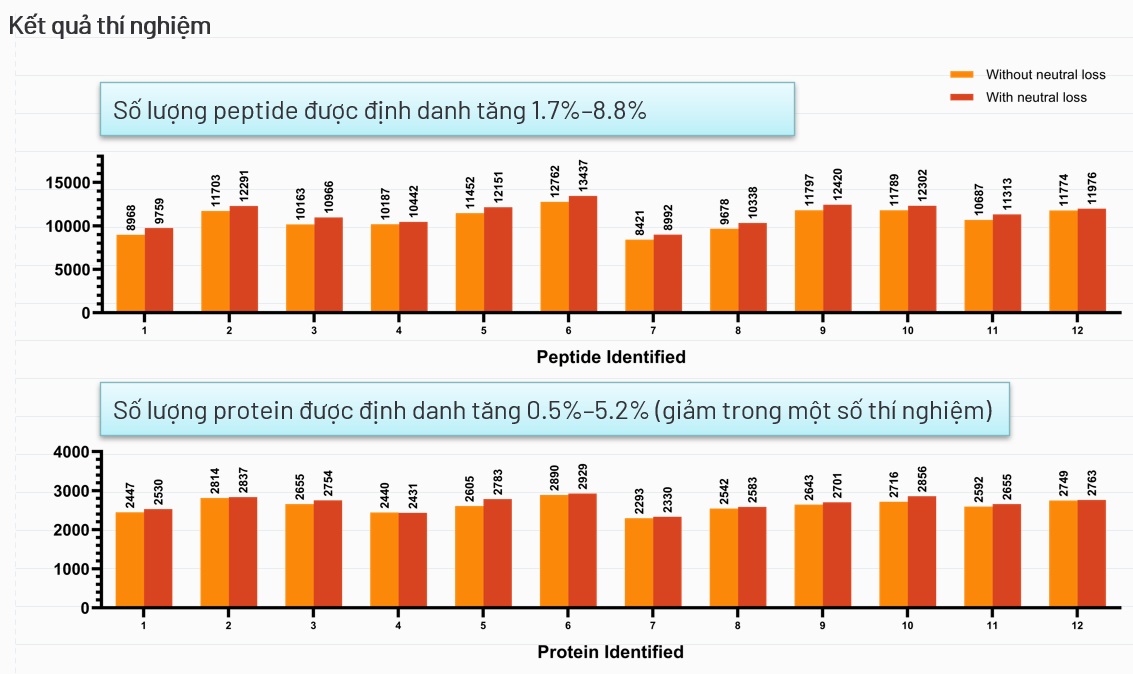
Chúng tôi sử dụng AlphaPeptDeep - một framework sử dụng học sâu dự đoán khối phổ rất tốt, để xây dựng khối phổ cho peptide (thay vì phải thực hiện thí nghiệm trên các máy phân tích khối phổ vật lý) và DIA-NN - một phần mềm giúp thực hiện phân tích DIA. Đầu tiên, chúng tôi xây dựng hai thư viện phổ trong đó một thư viện có thông tin neutral loss và thư viện phổ còn lại không chứa thông tin neutral loss. Hai thư viện phổ này khi phân tích với DIA-NN sẽ được dùng để so sánh về lượng peptide và protein được định danh. Bộ dữ liệu được sử dụng là PXD010595 từ trang PRIDE - nguồn trang uy tín về dữ liệu sinh học. Kết quả trên bộ dữ liệu này cho thấy thư viện phổ chứa thông tin neutral loss có số lượng peptide được định danh tăng từ 1.7% đến 8.8% và số lượng protein được định danh tăng từ 0.5% đến 5.2% so với thư viện không chứa thông tin neutral loss. Kết quả này không chỉ làm rõ ảnh hưởng của neutral loss mà còn chứng minh tiềm năng của các mô hình học máy và học sâu trong việc giải quyết thách thức này. Nghiên cứu cũng đặt nền tảng cho các nghiên cứu khác về neutral loss trong tương lai, giúp cải thiện khả năng phân tích protein và peptide trong sinh học phân tử.
Từ khóa: proteomics, phosphoproteomics, mass spectrometry, DIA, DDA, neutral loss
GitHub:
https://github.com/KhangSimple/DATN
https://github.com/digitalbiologylab/aiproteomics-phospho-neutral-loss